几种基于氨基酸序列构建很简单蛋白质三维结构的工具
几种基于氨基酸序列构建很简单蛋白质三维结构的工具
Several tools for constructing three-dimensional structures of very simple proteins based on amino acid sequences
文/Sobereva@北京科音 2023-Oct-15
经常有人问已知很简单蛋白质(小肽)的氨基酸序列,怎么得到其三维立体结构,这里就介绍几种工具。本文并不是对相关程序做完整的收集,介绍的这些已经足够满足各种实际情况的需要了。注意本文的范畴和蛋白质结构预测完全不同,那类程序是用于构造有三级结构的较复杂蛋白质用的,本文涉及的工具顶多只牵扯到二级结构。
需要注意的是,只有其中部分程序可以产生原子名符合IUPAC规范的pdb结构文件。仅对于这种情况,pdb文件才可以载入VMD后正常显示二级结构(至少得骨架重原子名满足IUPAC规范),以及能够直接被比如GROMACS的pdb2gmx所处理来产生拓扑文件。
笔者另外写了篇文章介绍通过输入序列产生DNA或RNA三维结构的工具,见《几种基于核酸序列构建三维结构的工具》(http://sobereva.com/692)。
1 ProBuilder
这是个在线程序,网址是https://www.ddl.unimi.it/vegaol/probuilder.htm。用户只需要输入氨基酸序列,比如RLCVVPIRLPPLRERT,并且指定要产生的二级结构的phi、psi、omega角度,就可以获得结构文件。能产生的格式很多,包括符合IUPAC命名规范的pdb文件。此程序产生的结构可以要求带和不带侧链,带侧链的情况由于不会自动做结构优化,因此侧链往往有严重重叠,后面介绍的很多程序也都是如此。
2 GaussView
Gaussian的官方界面程序GaussView提供了生物分子片段库,如下所示。可以选择位于链中间、N端、C端的各种氨基酸残基片段,在建模窗口点击一次出现第一个残基后,再一次一次点击肽键末端,就可以逐渐得到满足要求的肽链。
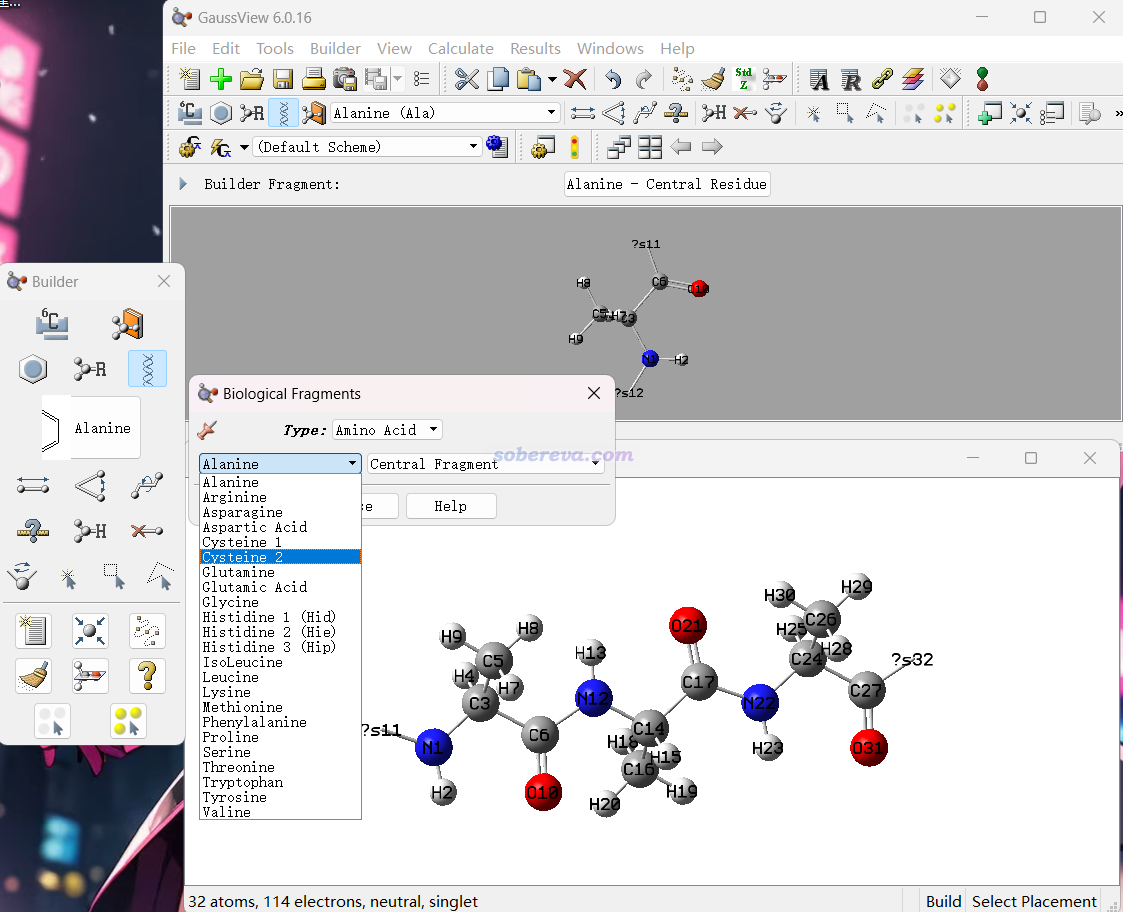
建好后可以保存成结构文件,包括pdb格式。用此程序的优点是可以自行精细修改三维结构,缺点是没法把一长串序列一下子转化为三维结构,而且这么保存的pdb文件里的原子名只是元素名而不是满足IUPAC规范的名字,里面连残基名都没有,而且也没法像ProBuilder那样直接设置对应特定二级结构的phi和psi角度(得手动一个一个调)。
3 Avogadro
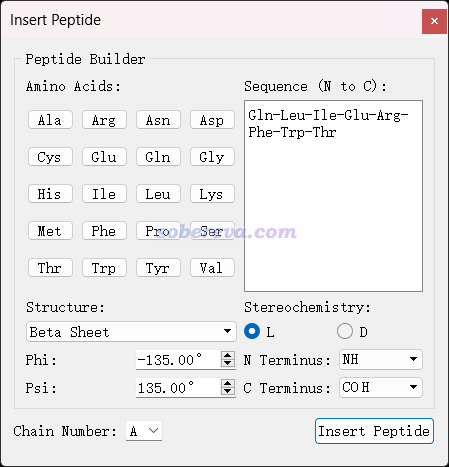
免费的可视化程序Avogadro(http://avogadro.cc)的Build - Insert - Peptide界面可以构建有特定二级结构、特定序列的小肽的三维结构,界面如下所示。可以保存出许多格式,保存成pdb的话原子名和残基名都是符合IUPAC规范的。此程序还自己支持GAFF、MMFF94等力场做几何优化,可以用于把建模后侧链的不合理接触消除掉。
4 gabedit
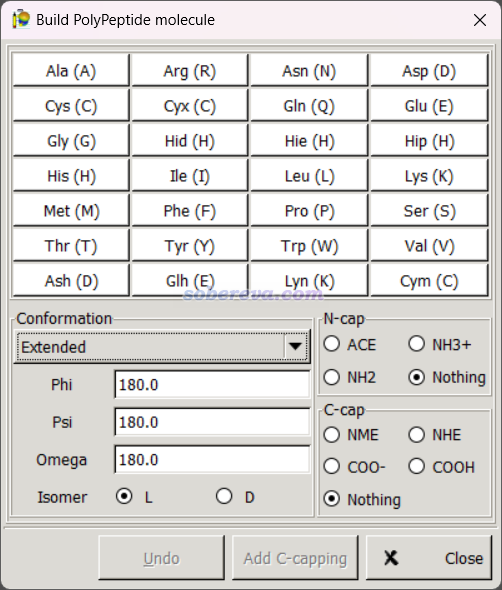
启动免费的可视化程序gabedit(http://gabedit.sourceforge.net)后,点菜单栏的Geometry - Draw,在新窗口里点右键,选Build - PolyPeptide,就可以看到构建多肽的界面,如下所示,也可以类似于Avogadro去产生符合特定二级结构的指定序列的小肽。每点击一次氨基酸的按钮在图形窗口里就会长出来相应的残基。之后点右键选save as,就可以保存成各种格式,包括满足IUPAC原子名和残基名规范的pdb格式。
5 AmberTools中的leap
知名的AmberTools(http://ambermd.org)程序里的leap可以用于根据氨基酸序列产生线型小肽的三维结构并保存成符合IUPAC命名规范的pdb文件。
参考手册或《Amber14安装方法》(http://sobereva.com/263)安装AmberTools后,在命令行窗口输入诸如tleap -f leaprc.protein.ff14SB即可启动纯文本界面的leap(tleap)且同时载入AMBER14SB力场相关的氨基酸的库文件。在里面可以输入比如如下命令定义一个序列,其中残基的N结尾和C开头分别代表N端和C端残基
TC5b = sequence { NASN LEU TYR ILE CGLN }
之后再运行以下命令就可以把序列以三维结构保存成/sob目录下的TC5b_linear.pdb了
savepdb TC5b /sob/TC5b_linear.pdb
6 PeptideBuilder
PeptideBuilder是个基于Python的轻量级构建小肽的库,网址是https://github.com/clauswilke/PeptideBuilder,具体介绍见原文PeerJ, 1, e80 (2013)。
在Linux下联网状态运行pip install PeptideBuilder即可安装。之后可以基于PeptideBuilder写Python脚本来构造小肽结构,可以指定psi和phi角度、有哪些残基等。此程序只产生重原子坐标,没有氢。产生的pdb文件符合IUPAC命名规范。
以下是个例子,比如保存为createQ6.py,之后运行python createQ6.py,就可以在当前目录下得到含有6个谷氨酰胺的yohane.pdb文件。
from PeptideBuilder import Geometry
import PeptideBuilder
geo = Geometry.geometry("Q")
geo.phi = -60
geo.psi_im1 = -40
structure = PeptideBuilder.initialize_res(geo)
for i in range(5):
PeptideBuilder.add_residue(structure, geo)
PeptideBuilder.add_terminal_OXT(structure)
import Bio.PDB
out = Bio.PDB.PDBIO()
out.set_structure(structure)
out.save("yohane.pdb")
有人基于PeptideBuilder做了扩展写了额外Python程序,能生成具有指定序列在N端加上Fmoc或ACE(乙酰)封闭、在C端加上NME(甲胺基)封闭的pdb文件。见《生成任意序列的封端短肽pdb的脚本CappedPeptideBuilder.py》(http://bbs.keinsci.com/thread-30278-1-1.html)。一个简单例子:先把PeptideBuilder装上,然后把CappedPeptideBuilder.py放在当前目录下,再运行python CappedPeptideBuilder.py -s KEYIN -p test,此时当前目录下就出现了test目录,其中ACE_KEYIN.pdb就是N端加了乙酰、C端加了甲氨基的序列为KEYIN的小肽结构了(没有氢)。
7 Tinker
动力学模拟程序Tinker有根据序列建立蛋白质三维结构的命令protein,参见计算化学公社论坛的帖子:http://bbs.keinsci.com/forum.php?mod=redirect&goto=findpost&ptid=11478&pid=79011&fromuid=1